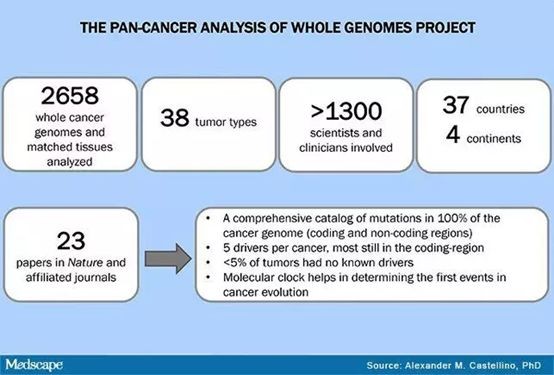
日前,“全基因组泛癌分析”联盟(PCAWG)对来自国际癌症基因组(ICGC)和美国癌症基因组图谱(TCGA)的38个癌种、超2600例原发肿瘤组织及其对应的正常组织进行全基因组测序和综合分析,发布了迄今最完整的癌症基因组图谱。研究结果于2月6日发表,其中6篇发表于Nature、15篇发表于Nature子刊。
普瑞基准联合创始人、MD安德森癌症中心梁晗教授参与了线粒体基因组相关工作,并在Nature Genetics发表了“人类癌症中线粒体基因组分子特征的全面描述”。线粒体是在癌症中起到关键作用的重要细胞器。研究分析PCAWG测序数据,得到了明确的线粒体基因突变图谱,并确定了一些超突变的案例。结果发现,在肾癌、结直肠癌和甲状腺癌中常出现线粒体基因的截短突变,提示这些信号通路的变化具有致癌作用。线粒体DNA存在频繁的核转移,其中一些会破坏靶向治疗。线粒体拷贝数变化在癌症内部和不同的癌症之间差异很大,且与临床变量相关。共表达分析突出了线粒体基因在氧化磷酸化,DNA修复和细胞周期中的功能,并显示它们与临床上的可干预基因位点存在关联。该研究为线粒体生物学意义转化为临床应用奠定了基础。

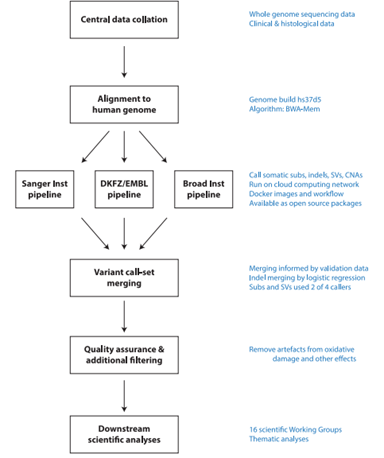
癌症由基因突变驱动的,大规模测序的出现可以在整个基因组范围内对这些突变进行系统的记录。PCAWG研究共纳入2658例癌症患者的肿瘤组织以及正常组织的全基因组测序结果,覆盖常见的38个癌种。该研究对于变异检出使用了统一的算法,变异检出标准和质控标准。检测突变共包括43,778,859个体细胞SNVs,410,123个体细胞多核苷酸变异,2,418,247个体细胞InDels,288,416个体细胞SVs,19,166个体细胞逆转录转位事件,8185个de novo线粒体DNA突变。这些突变根据肿瘤突变负荷和肿瘤种类的不同而有异质性。很多癌症中还存在复杂的染色体重排(17.8%)和染色体碎裂(22.3%)。
普瑞基准发布迄今最完整的癌症基因组图谱鉴于目前的认识,这些突变推动了超过2500个肿瘤的发生。研究者发现,91%的肿瘤至少有一个已经识别的驱动突变,平均每个肿瘤鉴定出4.6个驱动突变。对于编码区的点突变,每个肿瘤约有2.6个驱动突变,这也与TCGA预测的癌症相关基因结果相似。为了了解非编码区驱动点突变的突变比率,研究者将非编码区域已知的启动子和增强子与PCAWG研究中新发现的启动子和增强子结合起来。使用这种方法,PCAWG中至少有13%的驱动点突变在非编码区域,PCAWG中25%的肿瘤至少有一个是非编码区驱动点突变。这些突变中有三分之一是影响TERT启动子的突变
总体来说,非编码区的驱动点突变比编码区的突变比率要低。此外,PCAWG所有肿瘤类型中,SVs和点突变对于肿瘤的驱动在不同的癌种中是不一样的。乳腺癌和卵巢腺癌中,SVs是最常见的突变类型。结直肠腺癌和成熟B细胞淋巴瘤中驱动点突变发生率比较高。 嫌色性肾细胞癌和胰腺神经内分泌肿瘤患者中没有找到驱动突变。不同的肿瘤类型,突变会影响不同的基因组元素。研究证实,许多影响肿瘤抑癌基因的突变是双重打击失活事件。